Diagnostyka molekularna przy użyciu metody PCR (reakcja łańcuchowa polimerazy) polega na powieleniu konkretnego fragmentu materiału genetycznego (DNA lub RNA).
Technika ta pozwala z dużą czułością i precyzją potwierdzić lub wykluczyć obecność chorobotwórczych patogenów w badanej próbce. Jest to szczególnie ważne w diagnostyce drobnoustrojów, których badanie metodami tradycyjnymi jest utrudniona np. diagnostyka Mycoplasm czy wirusów.
Nasze laboratorium posiada specjalistyczne wyposażenie umożliwiające wykonanie badań zarówno w technice Real Time PCR jak i PCR konwencjonalny. Do badań wykorzystujemy gotowe i zwalidowane zestawy odczynników firmy Kylt umożliwiające diagnostykę molekularną chorób bakteryjnych, wirusowych i pasożytniczych.
Oferta badań
- Bakterie
- Wirusy
- Pasożyty
- Obecność pałeczek Salmonella spp.
- Obecność drobnoustrojów Mycoplasma synoviae - MS
- Obecność drobnoustrojów Mycoplasma gallisepticum - MG
- Różnicowanie szczepów szczepionkowych (MSH) oraz terenowych drobnoustrojów Mycoplasma synoviae – MS-H DIVA
- Obecność drobnoustrojów Ornitobacterium rhinotracheale – ORT
- Obecność drobnoustrojów Avibacterium paragallinarum (Coryza)
- Obecność drobnoustrojów Campylobacter spp.
- Identyfikacja czynników wirulencji Escherichia coli – APEC
- Identyfikacja genów toksyn Clostridium perfringens
- Obecność wirusa wywołującego zapalenie krtani i tchawicy - ILT
- Obecność Adenowirusa grupy 1 – FADV-1
- Obecność drobiowego Metapneumowirusa podtyp A i B – TRT A&B
- Obecność drobiowego Paramyxowirusa – PMV-1, NDV
- Obecność Reowirusa – REO
- Obecność wirusa choroby Gumboro serotyp 1 – IBDV
- Obecność kurzego astrowirusa – CastV
- Obecność indyczego astrowirusa typ 1 i 2 – TastV
- Obecność astrowirusa zapalenia nerek – ANV
- Obecność Rotawirusa A i D – Rota A&D
- Obecność wirusa zapalenia oskrzeli (Avian Coronavirus) - IBV
- Obecność wirusa IBV oraz jego wariantów – IB panel
- Obecność wariantu 4/91 wirusa IB – IB-4/91 (IB-793B)
- Obecność wariantu Israel02 wirusa IB – IB-Israel02 (IB-Variant02)
- Obecność wariantu Arkansas wirusa IB – IB-Arkansas
- Obecność wariantu D1466 wirusa IB – IB-D1466
- Obecność wariantu D274 wirusa IB – IB-D274
- Obecność wariantu IB-80 wirusa IB – IB-80
- Obecność wariantu Italy02 wirusa IB – IB-Italy02
- Obecność wariantu Massachusetts wirusa IB – IB-Mass
- Obecność wariantu Q1 wirusa IB – IB-Q1
- Obecność wariantu QX wirusa IB – IB-QX (IB-D388)
- Obecność wirusa choroby Mareka – MDV
- Różnicowanie szczepu terenowego od szczepu szczepionkowego Rispens CVI988 choroby Mareka – MDV Rispens
- Obecność pasożytów Histomonas meleagridis
W związku z elastycznym systemem pracy w naszym laboratorium jesteśmy otwarci na indywidualną współpracę w zakresie nowych kierunków badań.
Materiał do badań
- narządy wewnętrzne
- wymazy z narządów wewnętrznych
- karty FTA
- pióra
- kurz z kurnika
- wymazy środowiskowe/czystościowe
- wyizolowane szczepy bakteryjne
Rodzaj pobranego materiału jest uzależniony od rodzaju szukanego patogenu.
Materiałem do badań mogą być:
| KIERUNEK BADANIA | POBIERANY MATERIAŁ |
|---|---|
| Reowirus | wątroba, migdałki jelitowe, stawy, śledziona, żołądek |
| Identyfikacja czynników wirulencji Escherichia coli APEC | serce, wątroba, śledziona, płuca, worki powietrzne, jajników, stawy, nerki, szpik kostny, mózg |
| Avibacterium paragallinarum | zatoki, tchawica, worki powietrzne |
| Adenowirus grupy 1 | wątroba, jelito, migdałki jelitowe, nerki, żołądek |
| Wirus zapalenia oskrzeli IBV | tchawica, płuco, migdałki jelitowe, jajnik, nerki |
| Zapalenie krtani i tchawicy ILT | tchawica, płuco |
| Gumboro IBDV | bursa Fabrycjusza, śledziona, tkanki limfoidalne |
| Mycoplasma synoviae | tchawica, płuca, zatoki, stawy, szpara podniebienna |
| Mycoplasma gallisepticum | tchawica, płuca, zatoki, szpara podniebienna |
| Geny toksyn Clostridium perfringens | Jelito |
| Paramyxowirus aPMV-1, NDV | tchawica, jelito, płuca, wątroba, śledziona, mózg |
| Ornithobacterium rhinotracheale | serce, zatoki, tchawica, płuca, worki powietrzne, stawy |
| Rotawirus | jelito |
| Choroba Mareka | pióra, żołądek, śledziona, wątroba, nerki, nerwy |
| Histomonas meleagridis | jelito ślepe, wątroba |
| Metapneumowirus podtyp A i B | Zatoki podniebienne, szpara podniebienna, tchawica, spojówka |
| Kurzy astrowirus CastV | Jelito, migdałki jelitowe |
| Indyczy astrowirus TastV | Jelito, migdałki jelitowe |
| Wirusowe zapalenie nerek ANV | nerki, migdałki jelitowe, jelito |
Prosimy o przesyłanie suchych wymazów, bez podłoży transportowych. W przypadku prób zbiorczych pulujemy maksymalnie 5 wymazów na jedno badanie. Zalecamy stosowanie wymazów szczoteczkowych (FloqSwabs, eSwabs itp.)
Instrukcja pobierania materiału na kartę FTA - Pobierz
Choroba Mareka
Prosimy zwracać uwagę aby pobrane pióra posiadały wilgotną dudkę. Można także przesyłać zabezpieczone pióra wraz z kawałkiem skóry, aby nie dopuścić do wysuszenia materiału.
Choroba Mareka, Histomonas meleagridis
Choroba Mareka, Salmonella spp.
ORT, APEC E. coli, C. perfringens, A. paragallinarum, Salmonella spp.
W celu ustalenia szczegółów oraz konsultacji prosimy o kontakt z laboratorium.
Metody badawcze
- PCR konwencjonalny
- Real-Time PCR
- Sekwencjonowanie

PCR czyli reakcja łańcuchowej polimerazy (z ang. Polymeraze Chain Reaction) polega na powielaniu określonej sekwencji DNA w warunkach laboratoryjnych. Składa się z wielokrotnie powtarzanego cyklu trzech etapów: denaturacja, przyłączanie starterów, elongacja. Dobranie odpowiednich starterów komplementarnych do fragmentów matrycy znajdujących się na obu końcach interesującego nas genu pozwala namnożyć szukaną sekwencję. Uzyskane produkty reakcji rozdzielane są w żelu agarozowym w polu elektrycznym a następnie uwidaczniane w świetle UV przy pomocy barwnika. Uzyskanie świecącego prążka na odpowiedniej wysokości potwierdza obecność szukanego materiału genetycznego w wyizolowanej próbce.
Kierunki wykonywane tradycyjną metodą PCR w naszym laboratorium:
- Avibacterium paragalinarum (Coryza)
- Adenowirus grupy 1
- indyczy astrowirus TastV
- Identyfikacja czynników wirulencji Escherichia coli – APEC
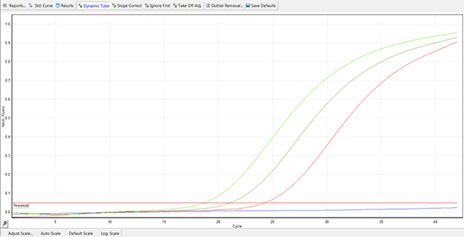
Real-Time PCR, czyli inaczej PCR w czasie rzeczywistym, zwanym również kinetyczną reakcją PCR lub ilościową reakcją PCR(ang. quantitive PCR). Nie należy jej mylić z RT-PCR (ang. Reverse Transcription).
Technika ta jest jedną z modyfikacji reakcji łańcuchowej polimerazy. Głównymi zaletami tej reakcji jest bardzo wysoka czułość reakcji, a także możliwość monitoringu ilości produktów reakcji po każdym jednym cyklu. Stosując różne barwniki fluorescencyjne możemy analizować jednocześnie kilka sekwencji DNA w jednej próbce. Znakowane nukleotydy bądź sondy umożliwiają wgląd w kinetykę reakcji, a co za tym idzie oszacowanie ilości produktu reakcji, co jest niemożliwe w konwencjonalnej metodzie PCR.
W przypadku próby pozytywnych podajemy wartość CT. Jest to moment wejścia reakcji w fazę logarytmicznego przyrostu ilości produktu. Im niższe CT tym większa początkowa ilość kopii badanej sekwencji w próbce.
W naszym laboratorium większość kierunków jest wykonywana metoda Real-Time PCR.
Informujemy, że nie wykonujemy oznaczania liczby kopii badanej sekwencji.
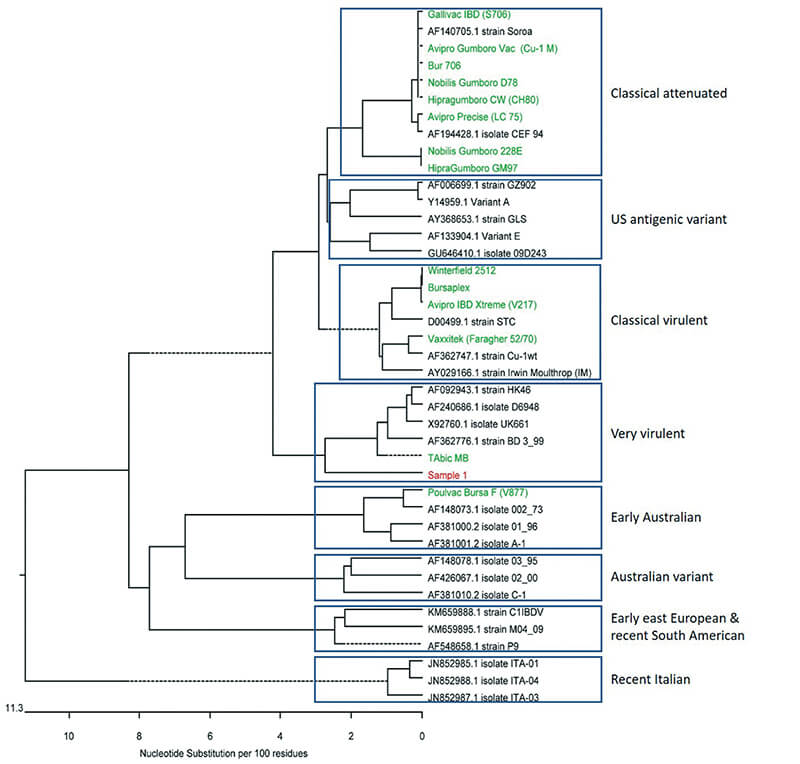
Sekwencjonowanie DNA to technika służąca do odczytywania kolejnych nukleotydów w cząsteczce DNA przy pomocy zautomatyzowanych sekwenatorów
Współpracujemy z renomowanym, międzynarodowym laboratorium w zakresie sekwencjonowania materiału genetycznego. Możliwość wykonania przykładowych badań:
- analiza filogenetyczna wirusa IBV
- sekwencjonowanie Adenowirusa
- sekwencjonowanie Reowirus
- sekwencjonowanie wirusa wywołujące chorobę Gumboro
- sekwencjonowanie astrowirusów
Kontakt

Jarosław Wilczyński Kierownik laboratorium